2024年7月11日
ポイント
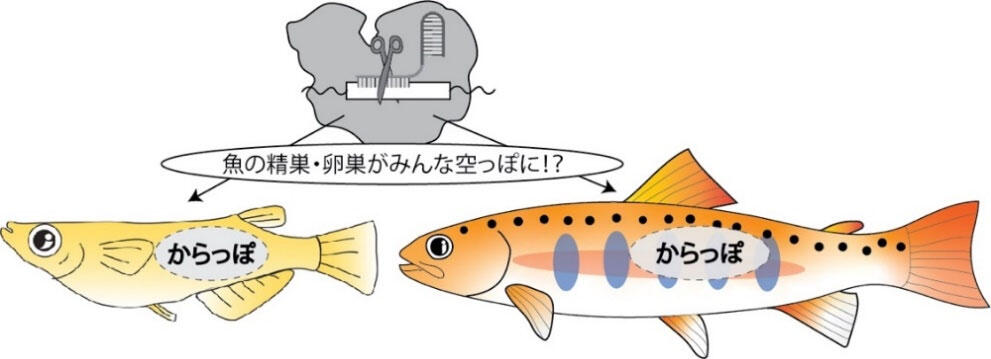
●CRISPR-Cas13dシステムを利用した汎用性の高い魚類の不妊化技術を開発。
●実験モデル魚であるメダカと産業重要魚種であるニジマスを高い効率で不妊化させることに成功。
●外来魚や養殖魚の迅速かつ確実な不妊化に期待。
概要
北海道大学大学院水産科学研究院の西村俊哉助教、藤本貴史教授、同大学北方生物圏フィールド科学センターの高橋英佑技術専門職員らの研究グループは、幅広い魚種で不妊化可能な技術の開発に成功しました。魚類の不妊化は、外来魚種の繁殖や遺伝子改変魚の逃亡による自然集団への遺伝子汚染を防ぐだけでなく、養殖魚の高成長・高肉質が期待できます。確実に魚を不妊化する方法として、精子と卵の元となる生殖細胞を欠損させ、精巣・卵巣を空っぽにする方法が有効ですが、幅広い魚類で汎用的に利用可能な不妊化技術は開発されていませんでした。
研究グループは、近年開発されたCRISPR-Cas13dシステムに着目しました。ゲノム編集技術として知られるCRISPR-Cas9システムでは、狙ったDNA(ゲノム)を切断し変異を導入できるのに対し、CRISPR-Cas13dシステムでは、狙ったRNAを切断し分解させることが可能です。標的となるRNAに結合するガイドRNA(gRNA)の設計に制約が小さいため、生殖細胞形成に必要な遺伝子の魚類共通配列を標的としたgRNAを設計することで、汎用的に魚類の不妊化が可能なのではないかと考えました。
そこで、ゲノムデータベース上に登録されている70魚種から取得したdnd1遺伝子(生殖細胞に必須の遺伝子)配列をアライメントし、配列共通性の高い領域を同定しました。残念ながら全魚種で100%一致する標的配列は無かったため、産業重要魚種や鑑賞魚(マグロ、サケ、ブリ、フグ、ティラピア、コイ、グッピー)、実験モデル魚(メダカ、ゼブラフィッシュ、トゲウオ)に代表される魚種グループの配列をもとにgRNAを設計しました。その結果、サケ専用gRNAをはじめ、マグロやブリ、フグ、ティラピア、コイ専用のgRNAでメダカを完全に不妊化できることが分かりました。さらにサケ専用gRNAによってニジマスの効率的な不妊化にも成功しました。
従来、魚種ごとに遺伝子のDNA配列を調べてからgRNAを設計する必要がありました。本研究で開発した共通gRNAは、全魚種の半分以上に適用できる可能性があります。そのため、本研究成果は、DNA配列が不明な外来魚や新規導入する養殖魚を迅速かつ確実に不妊化できる技術につながります。
なお、本研究成果は、2024年6月26日(水)公開のAquaculture誌に掲載されました。
論文名:Sterilization of fish through adaptable gRNAs targeting dnd1 using CRISPR-Cas13d System(CRISPR-Cas13dシステムにおけるdnd1遺伝子に対する共通gRNAを用いた魚類の不妊化)
URL:https://doi.org/10.1016/j.aquaculture.2024.741269
詳細はこちら